星标“医工学人”,第一时间获取医工交叉领域新闻动态~

当我们在谷歌中输入关键词,零点几秒内就能得到来自全球互联网的答案。但如果你的关键词是一段DNA序列,如何在存储着地球生命多样性密码的千万亿级(Petabase, PB)数据库中寻找它的踪迹呢?10月8日,苏黎世联邦理工学院的科学家们在《Nature》上发表文章,他们已经攻克了这一难题,成功构建了一个名为MetaGraph的“生物序列谷歌”,让检索全球生命数据变得前所未有的高效、廉价和精准。

在过去的二十年里,高通量测序技术的飞速发展将生命科学带入了一个“大数据”时代。从病毒、细菌到人类,从健康组织到癌变细胞,全球的实验室每天都在产生海量的基因、RNA和蛋白质序列数据。这些数据被存储在欧洲核苷酸档案馆(ENA)、美国国家生物技术信息中心(NCBI)等公共数据库中,其增长速度甚至超过了摩尔定律,目前总量已超过67 PB(6.7亿亿碱基对),形成了一片深不见底的“数字海洋”。
然而,拥有数据并不等于拥有知识。这片海洋虽然蕴藏着解开生命奥秘、攻克人类疾病的钥匙,但由于缺乏强大的导航工具,它在很大程度上仍是“只读”的。科学家们无法简单地“搜索”它。“想象一下,你想知道一种新发现的病毒序列是否曾在过去某个城市的地铁样本中出现过,”该研究的通讯作者之一André Kahles教授解释道,“在MetaGraph出现之前,你可能需要下载并分析数TB甚至PB的原始数据,这是一个极其消耗资源和时间的过程,对于大多数研究机构来说是不现实的。”
研究的核心是利用“带注释的德布鲁因图”(annotated de Bruijn graphs)数据结构。这并非一个全新的概念,但MetaGraph团队将其性能和规模推向了极致。其核心思想是将每一条长序列(如一个基因组)分解成数十亿个微小的、相互重叠的片段(k-mers),就像把一本书拆成无数个短语。然后,算法会识别出所有完全相同的“短语”,并将它们合并为图上的一个节点。这些节点再根据它们在原始序列中的前后关系连接起来,形成一张巨大的、错综复杂的网络图。
这个过程的魔力在于巨大的数据压缩。在包含数百万人类样本的RNA数据库中,由于个体间的序列高度相似,这种方法实现了高达7400倍的压缩。研究团队估计,一个容纳全球所有公共序列数据的完整MetaGraph索引,大小仅为170-230 TB,可以轻松存放在一个小型服务器机柜里,硬件成本仅需2500美元。一个曾经需要庞大计算集群才能处理的数据海洋,如今被浓缩到了几块硬盘之中。
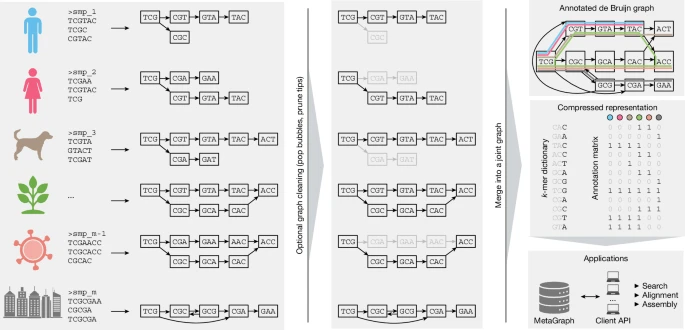
图1:MetaGraph框架。图示展示了图的构建与表示流程。单个测序样本(左侧)被组装成图(样本图),随后经过清理以去除虚假路径和错误组装(中部)。接着,样本图被合并形成MetaGraph索引(右上),该索引由压缩的k-mer字典索引和压缩的注释矩阵组成(右中;灰色部分表示可高效压缩且无需显式存储的内容)。该索引随后被用作下游应用的基础,例如序列搜索、组装及其他查询(右下)
压缩只是第一步,MetaGraph真正的颠覆性在于它极低的搜索成本。
通过创新的算法和云架构,研究团队证明,即使是面对全球所有公开的序列数据,进行一次小型查询(如搜索一个基因或一条病毒RNA)的成本也仅在100美元左右。对于需要比对数百万条序列的大规模研究,成本更是可以低至每兆碱基对0.74美元。
“这彻底改变了游戏规则,”《Nature》同期发表的新闻评论文章《‘Google for DNA’ brings order to biology’s big data》中引述一位同行学者的话说,“它将曾经遥不可及的全球数据分析能力,带给了每一个普通的生物实验室。”
为了证明MetaGraph的威力,研究团队展示了几个激动人心的应用案例。
他们利用MetaGraph索引的超过24万份人类肠道微生物组样本,分析了抗生素耐药基因(AMR)和噬菌体(一类感染细菌的病毒)在全球范围内的关联和演化趋势。分析发现,特定噬菌体的存在与某些耐药基因(如大肠杆菌中的β-内酰胺酶基因)的出现高度相关。他们还揭示了不同大洲耐药性随时间增长的显著趋势,例如在南美洲,针对“最后手段”抗生素替加环素的耐药性正显著增强。这种规模的实时监测,对于全球公共卫生预警具有不可估量的价值。
在另一项研究中,团队将目光投向了癌症。他们利用MetaGraph索引了来自癌症基因组图谱(TCGA)和GTEx项目的数万份人类RNA测序样本,去寻找一种难以被传统线性比对工具发现的特殊RNA分子——环状RNA(circRNA)。通过简单的图搜索,他们高效地识别出了数千个候选的环状RNA,并发现它们在不同癌症类型和正常组织中的表达模式存在显著差异,暗示了其在癌症发生发展中的潜在作用。
MetaGraph的出现,不仅仅是创造了一个搜索工具。它所构建的高度结构化的生物序列知识库,为未来更宏大的目标铺平了道路。研究者在论文中展望,MetaGraph可以作为一个高效的“数据引擎”,为训练面向生物学的AI大语言模型(LLMs)提供海量的、高质量的序列数据。
目前,研究团队已经将他们建立的部分索引通过一个名为“MetaGraph Online”的网站向公众开放,并提供了API接口。他们的最终愿景是,ENA和NCBI等大型数据存储库能够直接集成这种技术,为全球所有存储的序列数据提供实时的搜索服务。
正如论文所言,几年前还被认为是极具挑战性的任务——索引和搜索数千个测序数据集,现在已经可以在一台现代笔记本电脑上轻松完成。MetaGraph正引领我们进入一个生物数据探索的新纪元,在这个纪元里,每一个生命密码都触手可及。
[1] Karasikov, M., Mustafa, H., Danciu, D. et al. Efficient and accurate search in petabase-scale sequence repositories. Nature (2025).
https://doi.org/10.1038/s41586-025-09603-w
[2] Dolgin, E. "'Google for DNA' brings order to biology's big data." Nature (2025).
https://doi.org/10.1038/d41586-025-03219-w
END
撰文 | 郝娅婷
排版 | 周宇茜
审核 | 医工学人理事会
扫码加入医工学人,进入综合及细分领域群聊,
参与线上线下交流活动

推荐阅读
Nat. Commun. | 悉尼大学新研究:1分钟适当高强度运动,健康收益竟等于8分钟缓和运动,你的智能手表该更新算法了!
点击关注医工学人
最新直播


本篇文章来源于微信公众号: 医工学人